高效预测几乎所有人类蛋白质结构,AlphaFold再登Nature,数据库全部免费开放
以下文章来源于ScienceAI ,作者ScienceAI
关注人工智能与其他前沿技术、基础学科的交叉研究与融合发展。
这次,AlphaFold 的预测结果几乎覆盖了人类所有的蛋白质结构,而且将免费开放给公众。这将是科学界的一笔宝贵财富。

生命所必需的每一次基础生物学活动几乎都是由蛋白质带来的。蛋白质参与创建细胞和组织并保持着它们的形状;构成维持生命所需化学反应的催化酶;充当分子工厂、转运工具和马达;充当细胞通讯的信号和接收器等等。
蛋白质由很多氨基酸长链组成,通过折叠成精确的 3D 结构来完成无数的任务。这些结构控制着它们与其它分子互动的方式,决定了其功能以及它在疾病中的功能紊乱程度。阐明蛋白质的结构是分子生物学的核心议题,更是治疗患者、拯救生命、改变生活的医学发展的核心。
在这一领域,DeepMind 的 AlphaFold 被寄予厚望。在上周发表在《自然》杂志上的一篇论文中,DeepMind 表示,AlphaFold 预测的蛋白质结构已经能达到原子水平的准确度。
如今,这一成果的最大回报已经到来:其中一个研究小组刚刚宣布,他们已经使用新开发的 AlphaFold 预测出了 35 万种蛋白质的结构,包括人类基因组所表达的约 2 万种蛋白质和其他 20 种生物学研究中常用模式生物(如大肠杆菌、酵母和果蝇)的蛋白质,是以前用实验方法解决的蛋白质数量的两倍多。该组织表示,未来,他们还将继续扩大预测的范围,将预测数量扩展至已编目的所有蛋白质,这大约要覆盖 1 亿个分子。
最重要的是,DeepMind 还与欧洲生物信息研究所(EMBL-EBI)合作建立了一个平台——AlphaFold DB(AlphaFold 蛋白质结构数据库),将他们的预测结果免费开放给公众。这将涵盖 98.5% 的几乎所有人类蛋白。

网站链接:https://alphafold.ebi.ac.uk/
DeepMind 创始人兼首席执行官 Demis Hassabis 博士说:「我们使用 AlphaFold 生成了人类蛋白质组最完整、最准确的图片。我们相信这是迄今为止人工智能对推进科学知识所做的最重要贡献,也是人工智能可以为社会带来的各种好处的一个很好的例证 。」
这项最新的研究成果于 7 月 22 日以「Highly accurate protein structure prediction for the human proteome」为题发表在《自然》杂志上。

论文链接:https://www.nature.com/articles/s41586-021-03828-1
研究发现,AlphaFold 能对人类蛋白质组 58% 的氨基酸的结构位置给出可信预测。其中,对 35.7% 的结构位置的预测达到了很高的置信度,是实验方法覆盖的结构数量的两倍。在蛋白水平上,AlphaFold 对 43.8% 的蛋白的至少四分之三的氨基酸序列给出了可信预测。预测信息将通过 EMBL-EBI 托管的公用数据库免费向公众开放。
清华大学生命科学学院的龚海鹏副教授告诉机器之心——ScienceAI,「这项工作影响非常深远,将在很大程度上改变相关领域的科研模式」,而且从事蛋白质功能和机理研究、分子动力学模拟和基于结构的药物开发、蛋白质结构解析的研究者都能从中受益。
龚海鹏副教授表示,对于计算化学领域和生物信息学领域的研究者来说,这项研究给出了很好的蛋白质初始结构信息,可以基于此进行分子动力学模拟以及计算机辅助药物设计等工作。研究蛋白质功能和机理的学者,也可以根据预测的结构解释实验中氨基酸残基突变对蛋白质功能的影响,有助于更深入和更准确地理解蛋白质的工作机理。目前结构解析团队用冷冻电镜法较容易建立低分辨率的电子密度图,用X射线晶体衍射法常常缺乏相位信息,这些缺陷都影响结构的解析;而该研究结果的开源,使得大部分实验室可以根据精度较低的实验数据建立较为合理的结构模型。
不过,芝加哥丰田计算技术研究所的许锦波教授指出:「AlphaFold 预测人类蛋白的覆盖率很高,达到了 98.5%,但在某些生物制药或疾病治疗领域,对蛋白质氨基酸结构精度需要达到原子级别,从这个角度看,AlphaFold 还有一定的提升空间。」
另一些研究人员也同意许教授的看法。自然资源部第三海洋研究所一名研究蛋白互作的研究人员表示,AlphaFold 是对实验结构生物学的一种补充技术,它可以加速现有的生物结构实验研究,节约数月的时间,但「预测毕竟是预测,还是需要实验证实」。
参与这项工作的 DeepMind 研究人员表示,「像AlphaFold 这样擅长理解蛋白质结构任务的网络架构的发展令人乐观,我们可以在相关问题上(预测复合物的结构、结合非蛋白质成分等)取得进展……大规模的准确结构预测将成为一种重要工具,让我们能从结构的角度解答新的科学问题,而 AlphaFold 的预测结果将帮助进一步阐明蛋白质的作用。」
机器学习加速蛋白质结构预测
解析蛋白质的结构可以为理解生物学过程提供重要信息,并有望指导药物研发。考虑到理解人类蛋白质组对健康和医药的重要性,研究人员付出了大量努力来确定这些蛋白质结构。虽然开展了数十年的研究攻关,但只有 35% 的人类蛋白质映射到蛋白质数据库(PDB)条目。利用实验方法解析结构需要跨越诸多十分耗时的障碍,因此,扩大蛋白质组覆盖面仍是一项艰巨挑战。
蛋白质结构预测通过快速、大规模地提供可操作的结构假设,有助于缩小这一差距。近年来,机器学习方法的出现,让蛋白质结构预测取得了实质性进展。
2020年12月,DeepMind 团队开发的 AlphaFold2 在国际蛋白质结构预测竞赛 CASP14 击败一众选手,实现了前所未有的结构预测精度,这破解了出现50年之久的蛋白质分子折叠问题,被称作结构生物学「革命性」的突破、蛋白质研究领域的里程碑。
上周,备受瞩目的 AlphaFold2 开源,它预测的蛋白质结构能达到原子水平的准确度。鉴于此,DeepMind 将 AlphaFold 应用于人类蛋白质组结构预测。
AlphaFold方法
AlphaFold 网络由两个主要阶段组成。第1阶段,以氨基酸序列和多序列对齐(MSA)作为输入。它的目标是学习一个丰富的「成对表示」。这种表示可提供「哪些残基对在 3D 空间中接近」的信息。第2阶段,使用第一阶段的表示法直接产生原子坐标;将每个残基作为一个单独的物体,预测放置每个残基所需的旋转和平移;最终组装成一个完整的结构链。这里可以生成一个基于网络中间层表示的三维结构。
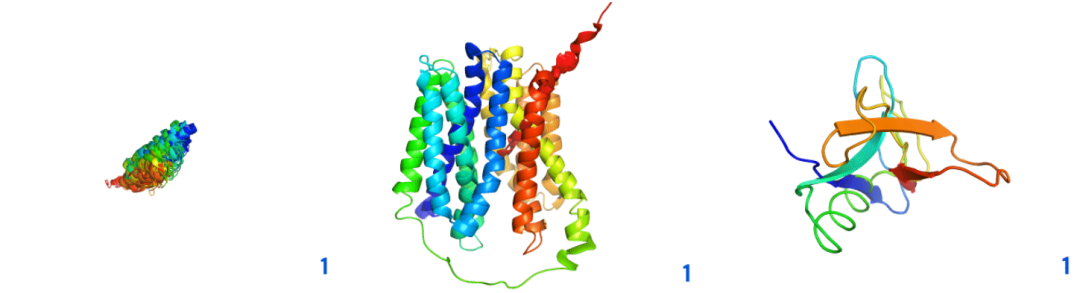
图示:CASP14的预测结构在网络的连续层上的目标T1044、T1024和T1064。(来源:论文)
准确性和置信度
CASP14 实验对 AlphaFold 进行了严格的评估。实验中,参与者对已经解决但尚未公开的蛋白质结构进行盲测。AlphaFold 在大多数情况下都取得了较高的精度,实验结构平均 95% 的 RMSD-Cα 小于1Å。在新发布的论文中,DeepMind在更大的 PDB 条目上进一步评估了这个模型。结果表明,AlphaFold 在大蛋白上具有强大性能和良好的侧链精度,其中主链预测性能很好。
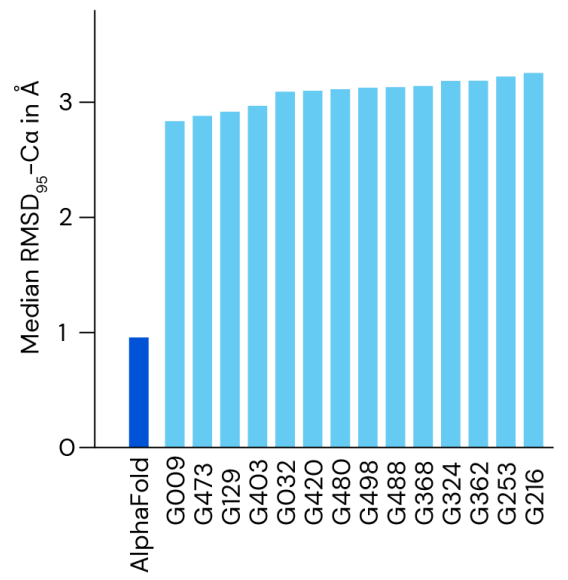
图示:在CASP14中AlphaFold相对于其他方法的精度。(来源:论文)
结构预测实用性的一个重要因素是相关置信度的质量。为了解决「模型能否确定其预测中可能可靠的部分」的问题,研究人员在 AlphaFold 网络的基础上开发了两个信任度量。
第一个度量是 pLDDT(预测的 lDDT-Cα),它是在 0 -100范围内对局部置信度的每个残基的度量。pLDDT可以沿着一条链显著变化,使得模型能够表达结构域的高置信度,但是在结构域之间的连接子(linker)上具有低置信度。研究人员提出了一些证据,证明低 pLDDT 的区域可能是孤立的非结构。pLDDT<50 的区域不应被解释,或者被解释为「可能的无序预测」。
第二个度量是 PAE(预测对齐误差),当预测和真实结构在残基y上对齐时,它报告AlphaFold在残基x处的预期位置误差。这对于评估对全局特征(尤其是域包装)的信心很有用。对于来自两个不同结构域的残基x和y,在(x,y)处始终较低的PAE表明α折叠对相对结构域位置更可信;在(x,y)处始终较高的PAE表明不应解释域的相对位置。用于生成PAE的一般方法,同样适用于预测各种基于叠加的度量,包括 TM-score 和 GDT。
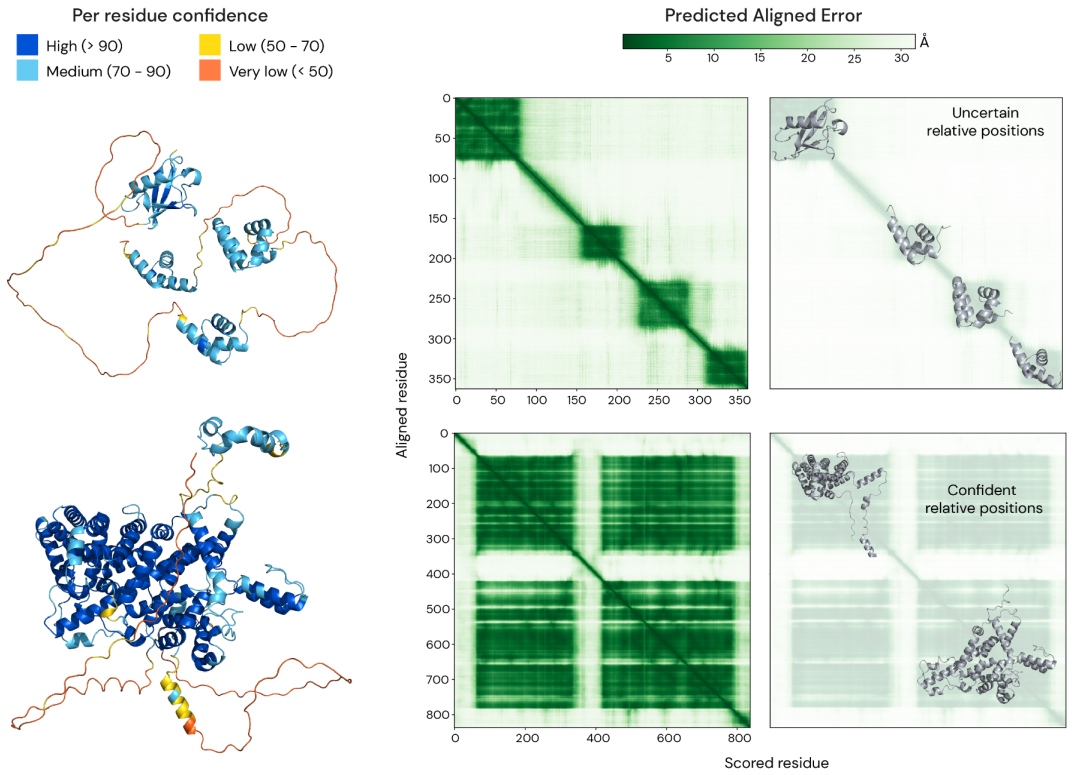
图示:两种示例蛋白(P54725、Q5VSL9)的每残基置信度(pLDDT)和预测对准误差(PAE)。(来源:论文)
蛋白质组规模和 AlphaFold DB
AlphaFold 可快速预测的特性,允许该方法应用于整个蛋白质组规模。研究人员使用 AlphaFold 对人类蛋白质组进行预测。在这之前,AlphaFold 已经对许多模式生物、病原体以及经济上重要的物种的参考蛋白质组进行了预测。观察物种之间pLDDT 分布的差异,AlphaFold 的预测对细菌和古细菌的置信度普遍较高,对真核生物的置信度较低;研究人员猜测可能与这些蛋白质组中疾病的流行有关。
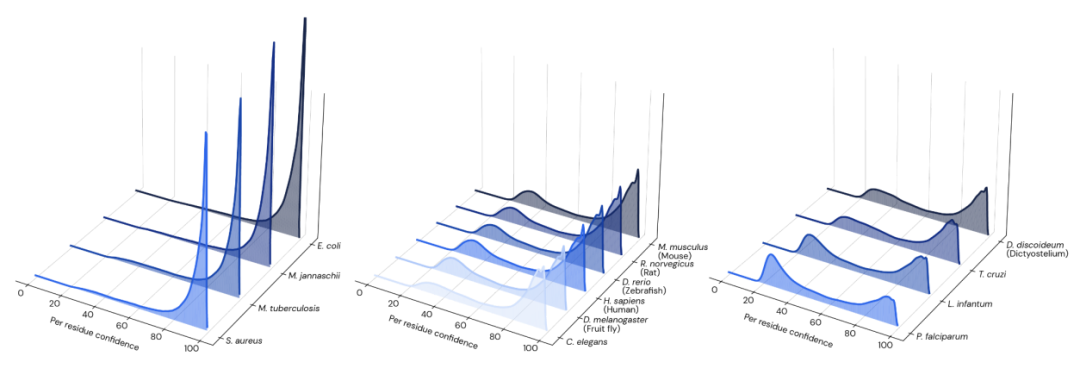
图示:14 个物种的每个残留置信度分布;从左到右:细菌/古细菌、动物和原生生物。(来源:论文)
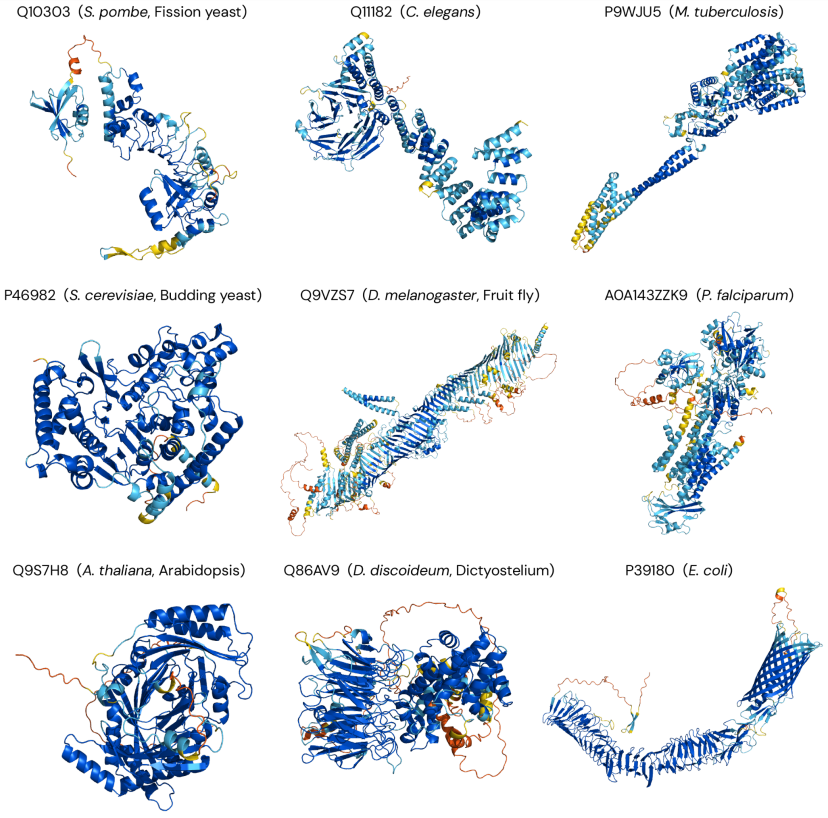
图示:AlphaFold DB 对自各种生物体的预测示例。(来源:论文)
论文一作 Kathryn Tunyasuvunakool 表示:「我们对计算生物学的未来感到兴奋。最终,我们希望 AlphaFold 将成为一个照亮蛋白质空间的有用工具,我们期待看到它在未来几个月和几年的应用。」
近期机器之心知识站将举行在线直播论坛,邀请领域专家就基于机器学习的蛋白质结构预测开展讨论。详情请关注后续报道,或添加机器之心小助手,注明「蛋白质」入群。

「2021 AI 100 Connect Webinar」AI + 工业专场

© THE END
转载请联系本公众号获得授权
投稿或寻求报道:content@jiqizhixin.com